基于PCR的测试方法是高度灵敏的,相比于VOA而言需要更少的血液。但是,在PCR测试方法中,人们通常针对单个保守性的靶点进行检测,这会极大地过高估计具有复制能力的HIV 病毒库的大小,这是因为大多数整合到宿主细胞DNA中的HIV前病毒都是缺陷性的,而且针对单靶点的PCR测试并不区分完整的HIV前病毒和具有DNA缺失和/或功能失活突变的HIV前病毒。可靠地估计遗传上完整的HIV前病毒需要验证HIV基因组上的多个区域的存在,以及所检测每个区域上的靶序列是否来自相同的HIV前病毒。
微滴式数字PCR(ddPCR)提供了一种替代性选择。在ddPCR中,PCR反应液(包括模板DNA)被分割成数千个单独的液滴,每个液滴的PCR检测结果分别报告。2019年报道的一种ddPCR测定方案利用了此类多重方法,在每个液滴内探测HIV-1基因组的两个区域。
在一项新的研究中,美国研究人员使用两种针对HIV基因组的3个区域(三重)的ddPCR测定方法(下称测定方法1和测定方法2)来开发一种5区域测试方法(这两种三重ddPCR测定各自针对两个独特的HIV基因组区域,但共同针对1个重叠区域,从而允许批间质量控制)。通过结合这两种平行的三重ddPCR测定方法,这些作者有信心对真正完整的HIV-1病毒基因组进行定量确定。作为进一步的改进,他们优化了其中的一种特异性定量确定T细胞的多重ddPCR测定方法,以便准确地将定量确定的数量与待研究的HIV靶细胞的数量进行正常化。这个额外的步骤对于组织活检特别有用,因为与血液相比,组织中的细胞群体难以分离和纯化。相关研究结果发表在2021年4月20日的Cell Reports Medicine期刊上,论文标题为“A highly multiplexed droplet digital PCR assay to measure the intact HIV-1 proviral reservoir”。

具体而言,测定方法1的三个HIV靶点位于HIV pol基因的3'端、tat基因和env基因,而测定方法2的三个HIV靶点位于长末端重复序列(LTR)/gag区域、pol基因的5'端和env。每种测定方法中,针对每个HIV靶点设计特异性的引物和探针。在每种测定方法的三个HIV靶点中,有两个使用相同的染料进行探针检测,但浓度不同,以便能够在荧光振幅的X/Y图上区分不同的HIV靶点。这使得这些作者能够对含有不同靶点组合的液滴进行定量确定。针对env的引物和探针在测定方法1和测定方法2中是相同的,在201个临床样本中,这两种测定方法的env性能几乎相同。此外,在这些临床样本中,五对引物/探针检测靶点的失败率极低:gag 0.5%;3' pol 1%;env 3.1%(两种测定方法均如此);tat 3.6%;5' pol 6.3%。
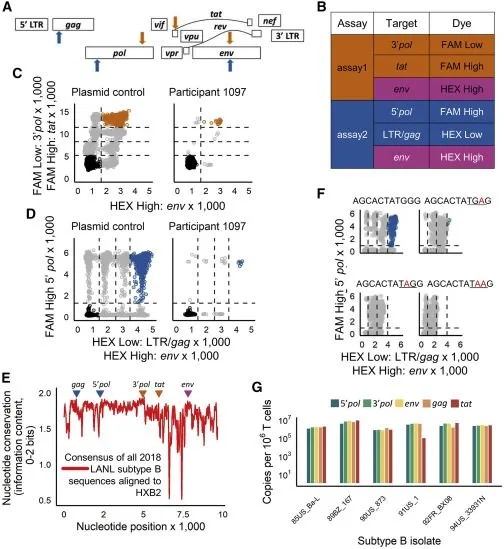
两种HIV-1三重ddPCR测定方法的设计,图片来自Cell Reports Medicine, 2021, doi:10.1016/j.xcrm.2021.100243
这些作者利用这种5区域测试方法评估了含有这5个区域的HIV前病毒的数量。他们评估的HIV前病毒数量平均比配对的定量VOA方法高12.1倍并且与后者相关(Spearman's ρ = 0.48),但是他们以此估计的HIV病毒库要比之前的DNA测定方法显著减少。
这些作者将他们的测试方法使用于来自接受抗逆转录病毒药物(ART)治疗的HIV感染者的纵向血液样本和粘膜样本。在这些患者中,完整的HIV前病毒在血液CD4+T细胞中的衰减速度快于有缺陷的HIV前病毒,而且完整的HIV前病毒在粘膜T细胞和循环T细胞中的频率相似。
参考资料:
Claire N. Levy et al. A highly multiplexed droplet digital PCR assay to measure the intact HIV-1 proviral reservoir. Cell Reports Medicine, 2021, doi:10.1016/j.xcrm.2021.100243.